【文献分享】Generating herbicide resistant and dwarf rice germplasms through precise sequence insertion or replacement
精确的植物序列插入或替换在技术上具有挑战性,但在作物育种中具有重要意义。已经得到开发的引导编辑(PrimeEditing)在植物中也有所应用并被优化,但对于有针对性的插入或替换较长的序列,效率仍然较低。除了PE编辑之外,类似在动物中已得到开发的编辑策略还有twinPE和GRAND编辑,具有一定的应用前景。
2023年11月22日,Plant Biotechnology Journal(IF: 13.8)在线发表了朱健康院士和王木桂副研究员团队的最新研究进展:“Generating herbicide resistant and dwarf rice germplasms through precise sequence insertion or replacement”。该研究比较了PE和GRAND编辑策略在水稻基因组中进行DNA片段插入、替换的效率,并利用GRAND策略在多个场景进行应用,成功替换、修复了多个水稻基因,展示了GRAND编辑在植物基因组精确和高效地插入或替换DNA片段的功能,为作物育种提供了新工具。
该团队发现,HPPD抑制剂除草剂如β-三酮类化合物,对已经出现的抗药杂草有效果,而水稻的HIS1基因赋予了对三酮类除草剂的广谱抗性,但在三酮类除草剂敏感的籼稻品种中(如籼稻S1035),发现了一个含有28碱基缺失、失活的HIS1等位基因。该等位基因导致水稻对三酮类化合物敏感,使得 HPPD抑制剂除草剂的应用会带来巨大风险。
因此他们尝试了PE和GRAND两种策略实现插入这28个bp以恢复HIS1基因的功能。
PE系统逆转录模板(RTT)中的与基因组的同源序列使得新转录的DNA与基因组DNA结合,形成双链DNA,然后,将在DNA修复机制下原基因组5ʹ端残基开始裂解,将3ʹ端链插入基因组。相反,如果RTT与靶区没有序列相似性,则3ʹ端链就不能与基因组序列杂交,新转录的DNA将被裂解。在David刘最初对PE系统的开发里,他们能用实现30~50bp的插入,PE3在动物细胞中能实现44bp的插入,效率达到20%。
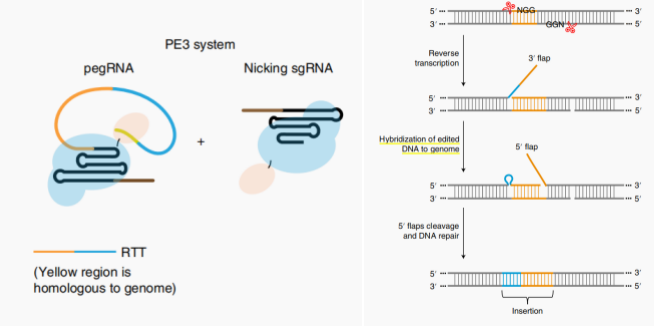
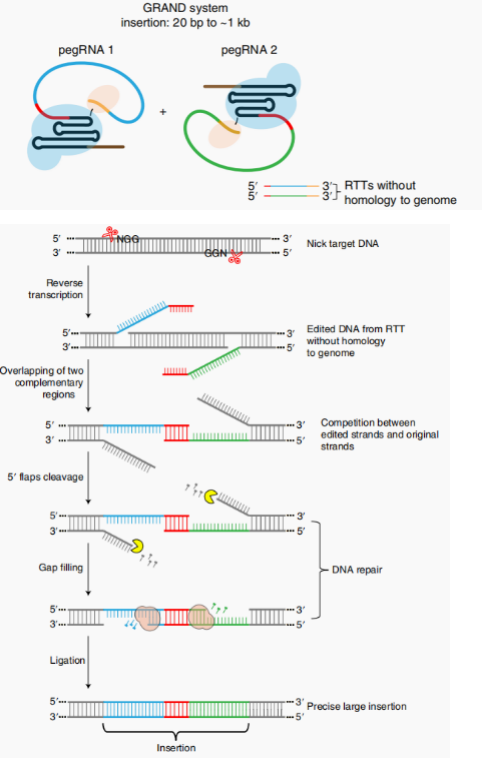
GRAND使用两个识别分别PAM序列的Cas9-RT分子分别结合并切割位置相反的目标DNA链。缺口位点的3ʹ端链与相应的pegRNA的PBS杂交,然后RT启动并扩展所需的两个新的单链dna(ssDNAs),它们通过互补端相互结合。3ʹ端链在DNA修复机制下被降解。因此,这一对pegRNA不需要与基因组序列有同源的RTT序列。在GRAND的初开发里,他们实现了在动物细胞插入150bp和250bp,效率分别为63.0%和28.4%。精准插入48bp,其中在两个切割位点间删除53bp后再插入101bp,效率达到42.7%。
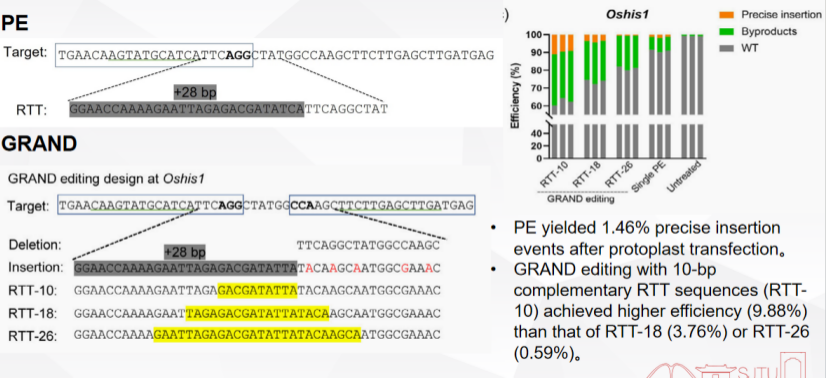
通过设计,该团队分别用PE和GRAND对水稻HIS1基因进行精准插入,原生质体转染后显示,PE的精确插入效率很低,仅为1.46%。而具有10个碱基互补RTT序列(RTT-10)的GRAND编辑效率(9.88%)高于RTT-18(3.76%)和RTT-26(0.59%)。
接着作者在遗传稳定转化过程中进一步评估GRAND编辑。作者用一种广泛使用的β-三酮类除草剂硝磺草酮60μM直接处理转基因系,最终RTT-10转化获得9株(11.5%)抗性系,而RTT-18和RTT-26转化仅各获得1株。在稳定转化得到的11个抗除草剂株系中,包含7个He型突变,即杂合子插入,所期望的插入只发生在一个等位基因上,而不管另一个等位基因的基因型如何;2个Bi型突变,即双等位基因插入,两个等位基因都包含所期望的插入和包含不同的无义突变,和2个Chi型突变,即包含精确插入事件的嵌合突变体。
可以看到RTT-10具有显著的优势,他们又选取了明辉86这一品种,这是一个HIS1存在28碱基缺失的三系品种,用最好的RTT-10编辑插入并稳定转化后,得到的84株转基因系中,有13株(15.5%)产生了对硝磺草酮的抗性。

该团队后续又对HIS1-RTT-10 #3的T1代进行鉴定外源TDNA鉴定,鉴定出了来自S1035编辑系的无T-DNA、纯合子后代。下右图是后代鉴定的PCR结果,a图鉴定潮霉素抗性基因,b图鉴定Cas蛋白基因,可以看出3、10、13、16、18都是无外源TDNA插入的纯合子后代。
插入后的HIS1基因的表达水平与野生型相当,且编辑植株对硝磺草酮的抗性与具有功能性HIS1基因的粳稻品种秀水134相似。这些结果表明,GRAND编辑可以用于救助其他有缺陷的品种,快速解决在水稻应用 HPPD抑制剂除草剂的风险。
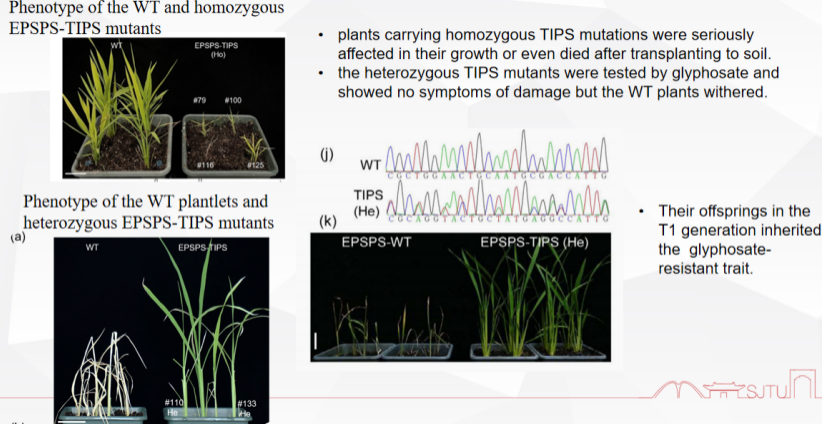
之后,该团队还在OsEPSPS基因中进一步测试PE和GRAND策略,即在OsEPSPS基因中导入耐草甘膦的TI/PS双点突变(C518T + C529T)。编辑设计思路和上一个基因的思路相似。原生质体测试显示,GRAND编辑的效率是PE的1.86倍(9.98% vs 5.37%)。PE仅有3株(2.0%)杂合子包含了预期的双点突变,其他突变体要么是嵌合体,要么是C518T和C529T独立发生。相比之下,GRAND编辑产生了5株(3.4%)纯合子和18株(12.2%)杂合子。
由于纯合TIPS突变植株在移栽土壤后生长严重受损或死亡,该团队测试了杂合TIPS突变体对草甘膦的反应。结果显示,杂合子没有损伤症状而野生型植株枯萎。这些后代都是GRAND编辑的后代。这些杂合子的T1后代遗传了耐草甘膦性状。
为了进一步评估GRAND编辑的活性,该团队又设计在OsSLR1基因的TVHYNP结构域替换一个含有3个氨基酸置换的28碱基片段。结果显示,在58株转基因系中,获得了4株(6.9%)纯合子和19株(32.8%)杂合子。如预期的那样,纯合子和杂合子都显示出生长矮化表型。
最后,作者总结了他们的工作,该研究测试了PE和GRAND编辑策略在水稻基因组中进行DNA片段的插入和替换效率,结果表明GRAND编辑的效率明显高于PE。且利用GRAND编辑成功修复了多个水稻品种中HIS1基因的28bp缺失,获得了对三酮类除草剂的抗性。还利用GRAND编辑在EPSPS基因中敲入了赋予对草甘膦抗性的点突变。GRAND编辑实现了在SLR1基因中替换28bp序列,获得了生长矮化表型。因此,该研究展示了GRAND编辑在植物基因组精确和高效地插入或替换DNA片段的功能,这为作物育种提供了新工具。


 loading......
loading......